모두가 양돈 현장에서 PRRS를 여러 차례 겪어 봤지만, 아직도 PRRS 컨트롤에 대한 '정답'은 없습니다. 오랜 기간 양돈농가를 괴롭혀온 만큼 PRRS에 대한 오해와 편견이 많이 쌓여있는 현실입니다. 'PRRS의 모든 지식'(총 15화)을 통해 우리 농장에 맞는 PRRS 컨트롤의 '해답'을 발견할 수 있길 기대합니다. 본 기고글은 HIPRA 본사에서 출간한 'The book for PRRS Knowledge"' 내용을 번역·정리한 것입니다.
유전적 다양성이 높은 PRRS 바이러스
초기 연구에서는 PRRS 바이러스의 유전형을 최초로 분리된 지역에 따라 유럽형(또는 Type 1, 대표 바이러스 Lelystad)과 북미형(또는 Type 2, 대표 바이러스 VR-2332)로 구분하였습니다. 이후 분리된 PRRS 바이러스들은 일반적으로 ORF5라는 가장 변이가 심한 유전자 부위를 기준으로 계통을 분류해왔습니다. PRRS 바이러스 유전자의 전체 염기서열 1만 5천 개에서 약 600개의 염기로 구성된 ORF5가 차지하는 비중은 4%에 불과합니다. 하지만 특징적인 변이를 볼 수 있다는 점에서 ORF5를 기준으로 분류하는 것이 일반적이며, 북미형과 유럽형의 두 유전형을 대표하는 바이러스 간 염기 서열 상동성은 55% 수준입니다.
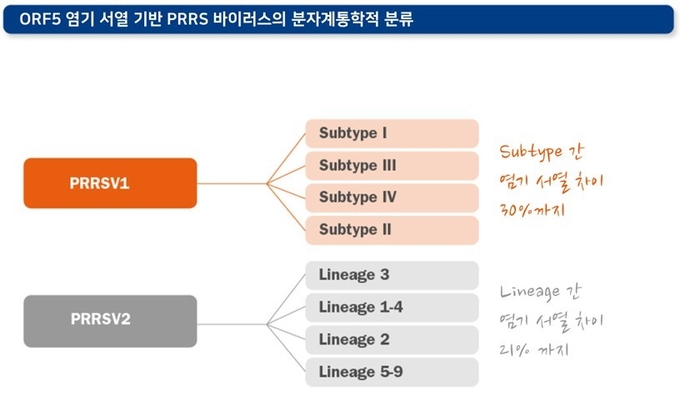
최근 바이러스 분류 국제위원회(ICTV, International Committee on Taxonomy of Viruses)는 이들을 두 개의 서로 다른 바이러스 종(species)인 PRRSV1과 PRRSV2로 규정하고 있습니다. 하지만 더 중요한 것은 같은 PRRSV1(유럽형)이나 PRRSV2(북미형) 내에서도 바이러스이 유전적 차이가 20% 이상으로 편차가 크다는 점입니다(PRRSV1에서 30%까지, PRRSV2에서 21%까지).
PRRS 바이러스는 변이가 심하고 높은 유전적 다양성을 갖고 있습니다. 두 개의 바이러스 종은 다양한 아형(subtypes)과 계통군(clades)으로 분류될 수 있습니다.
PRRSV1(유럽형)의 경우, 최소 4개의 아형(subtypes, 서브타입)이 존재합니다. 서브타입 I의 경우 서유럽 지역에서 우세하게 검출되며, 서브타입 II-IV는 폴란드 동부 국가들에서만 분리되고 있습니다. 서브타입 II-IV 내에서의 유전적 차이는 서브타입 I보다 더 큰 것으로 보입니다(ORF5 기준 각각 18.2% vs 11.9%).
PRRSV2(북미형)의 경우 서브타입이 명확하게 구분되지 않으며, 대신 두 개의 계통군(clades)에 속하는 여러 개의 계통(lineages, 리니지)으로 분류됩니다. 북미 지역 분리주들은 일곱 가지의 리니지로 나뉘며, 또 다른 두 가지 리니지는 동남아시아 지역에서만 분리되기도 합니다.
유전적 변이율은 PRRS 바이러스의 유전체 부위별로 다르게 나타납니다(ORF5뿐 아니라 ORF1a, ORF3, ORF4 부위 역시 높은 변이율을 보임). 앞서 설명한 대로 PRRS 바이러스의 아형(subtypes)이나 계통(lineages)을 결정하는 분석 방법(phylogenetic analyses)은 변이율이 높은 ORF5를 기반으로 이루어집니다. 하지만 ORF5가 전체 유전체 중 이 부분이 차지하는 부분은 4% 정도로 아주 작습니다. 전체 유전체의 다른 부위에서 일어나는 변이를 고려할 때, 현재의 ORF5 기반 분류법 역시 향후 바뀌게 될 가능성도 고려해볼 수 있습니다.
PRRS 바이러스의 유전적 다양성은 어떻게 나타날까?
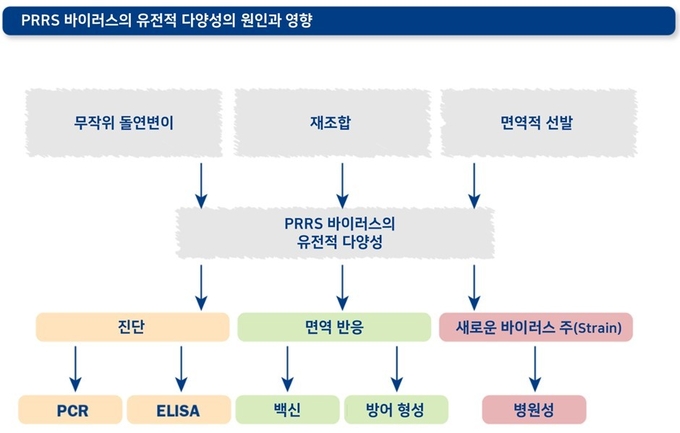
PRRS 바이러스의 유전적 다양성은 돌연변이(mutation), 재조합(recombination), 선발(selection)의 세가지 현상으로 나타나는 결과입니다.
● PRRS 바이러스의 무작위 돌연변이(Random mutation)
PRRS 바이러스의 변이 속도는 잘 알려진 조류 독감 바이러스(Avian influenza virus)나 인체 면역결핍 바이러스(Human immunodeficiency virus)의 40배 이상에 달할 정도로 빠르게 변이되는 바이러스입니다. 다른 RNA 바이러스처럼 PRRS 바이러스의 RNA 중합 효소는 RNA의 전사(transcription) 과정 중 발생하는 오류를 수정하는 능력이 없습니다.
PRRS 바이러스 유전자의 복제 과정 중의 오류는 100~1,000개의 염기를 읽을 때마다 발생하는데, 이러한 오류로 인해 복제된 바이러스는 이전과 다른 유전자 염기서열을 가질 수 있습니다. 결과적으로 기존의 부모 바이러스와 염기 서열이 다른 새로운 PRRS 바이러스들이 탄생하게 됩니다.
장기간 PRRS 바이러스에 감염된 돼지는 이처럼 유사한 변이 과정을 거친 수많은 변종 바이러스가 운집한 상태입니다. 감염된 돼지 체내에서 PRRS 바이러스의 다양한 변이 바이러스가 경쟁하면서 두드러지게 우점하는 바이러스가 나타나고 전체적인 감염의 특징을 결정짓게 됩니다. 이렇게 운집한 바이러스 집단을 변이가 매우 활발한 바이러스의 유사 변종 집단이라는 의미로 'Quasispecies'라고 부르기도 하고 일명 '변종구름(mutant clouds)'으로 칭하기도 합니다. 이는 각 바이러스 종(species) 및 숙주에서 독립적으로 발생하는 현상입니다. Quasispecies와 면역 억제로 인해 PRRS 바이러스가 돼지에서 만성적으로 지속 감염될 수 있다고 추정하고 있습니다.
● PRRS 바이러스 사이의 재조합(Recombination)
염기 서열의 차이가 매우 큰 PRRS 변이주의 경우 변이주가 단일 바이러스의 돌연변이 현상으로 생성되었다고 설명하기는 어렵습니다. 단순 돌연변이 만으로 단기간에 염기 서열에 큰 차이를 발생시키기 어렵기 때문입니다. 이를 보다 잘 설명할 수 있는 개념이 바로 바이러스의 재조합(recombination)입니다.
재조합은 두 개 이상의 PRRS 바이러스 주(strain) 사이에 유전 물질이 교환되는 것을 의미합니다. 이러한 재조합 여부를 확인하려면 PRRS 바이러스의 ORF5 부분만이 아닌 전체 염기 서열을 분석이 필요합니다. 전체 염기 서열을 시퀀싱을 통해 비교해보면, 실제로 ORF5의 유전정보는 동일하지만 서로 다른 바이러스가 재조합된 경우를 자주 확인할 수 있습니다. 이러한 결과를 통해 재조합 현상이 PRRS 바이러스의 유전적 다양성을 유발하는 매우 중요한 요인임을 유추할 수 있습니다.
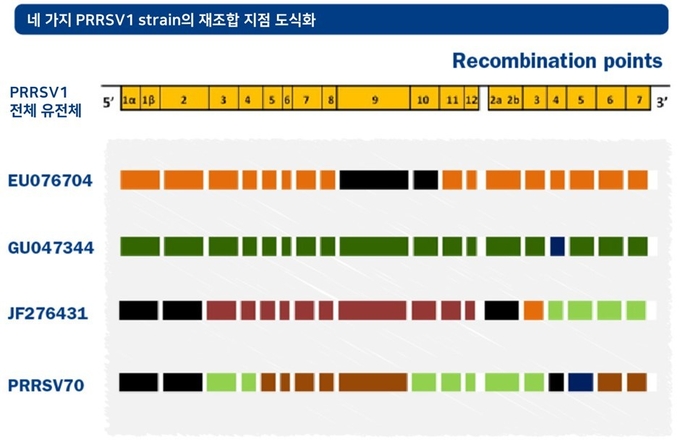
PRRS 바이러스 사이에서 재조합이 일어나는 경우 새롭게 탄생한 바이러스의 병원성, 증식 속도, 조직 친화성, 면역원성이 얼마나 나타날지 예측할 수 없습니다. 부모 바이러스들과 비교하여 재조합 바이러스의 특성을 예측하는 것도 불가능합니다. 중요한 것은 야외주와 백신주 사이에서 재조합이 발생할 수 있고, PRRSV1(유럽형)과 PRRSV2(북미형) 사이에서의 재조합도 가능하다는 점입니다. 예측하기 어려운 재조합 변이주의 발생을 줄이기 위해서는 접종 후 돈군 내 순환이 적은 백신을 선택하고 백신 접종 시기를 신중히 결정해야 합니다.
● 면역적 선발과정에서 나타나는 우점 바이러스
변이 바이러스가 혼재하는 Quasispecies 상태이거나 여러 바이러스에 동시에 감염된 상태의 돼지에서는 면역 회피 능력이 큰 쪽으로 변이된 바이러스가 우세한 변이주로 자리잡게 됩니다. 즉, PRRS 바이러스는 끊임없이 변이주를 생성하고 돼지의 면역시스템은 이에 대응하면서, 회피 능력이 큰 PRRS 변이주가 선발되어 우점하게 되는 것입니다.
짧은 간격으로 돈군을 모니터링하여 서로 다른 PRRS 바이러스들이 검출되었다면, ①외부로부터 신규 바이러스가 유입되었을 가능성과 ②기존 바이러스들 사이에서 우세 변이주가교체 되었을 가능성을 모두 고려해야 합니다. 정확한 판단을 위해서는 농장에서 검출된 바이러스에 대해 주기적으로 시퀀싱을 실시해 염기 서열 정보를 축적하는 것이 필요합니다.
PRRS 바이러스의 유전적 다양성이 가져오는 결과는?
유전적 변이가 빠른 PRRS 바이러스의 특성은 진단, 면역 반응, 새로운 바이러스 주(strain)의 등장, 병원성, 세포 친화성, 역학 등 많은 영역에 영향을 미칩니다.
● 유전적 다양성으로 인한 진단 시 유의사항
- ELISA : PRRSV1(유럽형)과 PRRSV2(북미형)에 대한 항체를 하나의 키트를 통해 검출할 수 있는 ELISA 키트가 많이 사용되고 있습니다. 한편 특수하게 디자인된 ELISA 키트를 활용하여 두 바이러스를 혈청학적으로 구분할 수 있도록 사용하는 경우도 있습니다. ELSIA 분석은 검사키트에 따라 특정 PRRS 바이러스에 대한 항체를 검출하지 못하는 위음성(False negative) 결과를 보이는 한계점도 갖고 있습니다. 여러 연구를 통해 이러한 위음성 현상이 밝혀지고 있으며, 특히 PRRSV1(유럽형)의 서브타입 II-IV에서 잘 나타나는 경향을 보이고 있습니다.
- PCR : PRRS 바이러스의 유전체에서 변이가 적은 보존적인 부위는 많지 않습니다. 점점 변이가 심해지는 PRRS 바이러스의 특성으로 인해, 항원 검출을 위한 PCR 디자인도 복잡해지고 있습니다. PCR의 타겟 부위로 이용되는 유전자 부위에 변이가 생기는 경우, 변이된 바이러스가 검출되지 않는 위음성 결과가 나타날 수 있으므로 주의해야 합니다.
● 예측할 수 없는 PRRS에 대한 면역반응
감염된 돼지에서 혼재하고 있는 PRRS 바이러스의 변이주들은 면역을 회피하는 방향으로 돌연변이를 만들어가며 지속 감염을 유발합니다. 이처럼 PRRS 바이러스의 빠른 변이율로 인해 재감염에 대한 방어능력은 부분적이거나 불완전할 수밖에 없습니다.
이러한 유전적 다양성으로 인해 모든 PRRS 바이러스에 대해 완전한 방어 능력을 갖춘 백신을 개발하기는 어렵습니다. 더불어 현재까지 PRRS 바이러스의 방어 면역에 관련된 유전자 정보가 무엇인지 명확하게 밝혀지지 않았습니다. 이러한 이유로 단순히 야외주와 백신주의 염기서열 상동성 비교만으로는 백신의 방어 효과를 예측하기 어려운 것이 현실입니다.
● 새로운 바이러스 주(strain)의 등장
PRRS 바이러스는 지속적인 변이를 통해 기존의 PRRS에 대한 편견을 깨는 방향으로 변화하고 있습니다. 지난 10여년 동안 아시아 지역의 일부 국가에서는 PRRSV2에 속하는 고병원성 PRRS 바이러스(HP-PRRSV)로 인한 큰 피해를 받아왔습니다.
국내에는 PRRSV1은 PRRSV2에 비해 병원성이 약하다고 생각하는 농장이 많지만, 병원성의 정도는 유전형에 따라 결정되는 것이 아닌 각 바이러스의 고유한 특징이라는 것이 다양한 사례들을 통해 증명되고 있습니다.
유럽에서는 PRRSV1인 Lena주를 비롯해 병원성이 높은 PRRS 바이러스가 확산하면서 경제적인 피해 규모가 커지고 있습니다. 벨라루스에서 처음 발생한 Lena주는 70%에 이르는 높은 폐사율과 고열, 전신적인 바이러스 증식, 강한 염증 반응을 일으키는 PRRSV1 강독주임이 밝혀졌습니다. 오스트리아의 농장들에서 분리된 AUT13-883과 AUT14-440 등의 바이러스 역시 뚜렷한 호흡기 증상과 심각한 폐 병변, 일당증체량 감소를 유발하는 PRRSV1으로 알려져 있습니다.
예측을 뛰어넘는 PRRS 바이러스의 변이 능력을 고려하면 앞으로 더 큰 피해를 유발하는 새로운 PRRS 바이러스가 등장할 수도 있습니다. 특히 안전성이 검증되지 않은 PRRS 바이러스를 직접 투여하거나 허가되지 않는 방식으로 백신을 사용할 경우 이러한 변이를 가속시킬 수 있습니다. 이러한 변이주 발생 위험성을 최소화하기 위해서는 안전성을 우선 순위에 두고 PRRS 백신을 선택해야 할 것입니다.

참고 문헌
-
Allende R, Laegreid WW, Kutish GF, Galeota JA, Wills RW, Osorio FA. Porcine reproductive and respiratory syndrome virus: description of persistence in individual pigs upon experimental infection. J Virol. 2000, 74:10834-7.
-
Allende R, Kutish GF, Laegreid W, Lu Z, Lewis TL, Rock DL, Friesen J, Galeota JA, Doster AR, Osorio FA. Mutations in the genome of porcine reproductive and respiratory syndrome virus responsible for the attenuation phenotype. Arch Virol. 2000, 145:1149-61.
-
Brockmeier SL, Loving CL, Palmer MV, Spear A, Nicholson TL, Faaberg KS, Lager KM. Comparison of Asian porcine high fever disease isolates of porcine reproductive and respiratory syndrome virus to United States isolates for their ability to cause disease and secondary bacterial infection in swine. Vet Microbiol. 2017, 203:6-17.
-
Chang CC, Yoon KJ, Zimmerman JJ, Harmon KM, Dixon PM, Dvorak CM, Murtaugh MP. Evolution of porcine reproductive and respiratory syndrome virus during sequential passages in pigs. J Virol. 2002, 76:4750-63.
-
Cortey M, Díaz I, Martín-Valls GE, Mateu E. Next-generation sequencing as a tool for the study of Porcine reproductive and respiratory syndrome virus (PRRSV) macro- and micro- molecular epidemiology. Vet Microbiol. 2017. doi: 10.1016/j.vetmic.2017.02.002.
-
Darwich L, Gimeno M Sibila M, Diaz I, de la Torre E, Dotti S, Kuzemtseva L, Martin M, Pujols J, Mateu E. Genetic and immunobiological diversities of porcine reproductive and respiratory syndrome genotype I strains. Vet Microbiol. 2011, 150:49-62.
-
Dea S, Gagnon CA, Mardassi H, Pirzadeh B, Rogan D. Current knowledge on the structural proteins of porcine reproductive and respiratory syndrome (PRRS) virus: comparison of the North American and European isolates. Arch Virol. 2000, 145:659-88.
-
Díaz I, Gimeno M, Darwich L, Navarro N, Kuzemtseva L, López S, Galindo I, Segalés J, Martín M, Pujols J, Mateu E. Characterization of homologous and heterologous adaptive immune responses in porcine reproductive and respiratory syndrome virus infection. Vet Res. 2012, 19:43:30.
-
Dokland T. The structural biology of PRRSV. Virus Res. 2010, 154:86-97.
-
Domingo E, Holland JJ. RNA virus mutations and fitness for survival. Annu Rev Microbiol. 1997, 51:151-78
-
Dunowska M1, Biggs PJ, Zheng T, Perrott MR. Identification of a novel nidovirus associated with a neurological disease of the Australian brushtail possum (Trichosurus vulpecula). Vet Microbiol. 2012, 156:418-24.
-
Frydas IS, Nauwynck HJ. Replication characteristics of eight virulent and two attenuated genotype 1 and 2 porcine reproductive and respiratory syndrome virus (PRRSV) strains in nasal mucosa explants. Vet Microbiol. 2016, 182:156-62.
-
Frydas IS, Trus I, Kvisgaard LK, Bonckaert C, Reddy VR, Li Y, Larsen LE, Nauwynck HJ. Different clinical, virological, serological and tissue tropism outcomes of two new and one old Belgian type 1 subtype 1 porcine reproductive and respiratory virus (PRRSV) isolates. Vet Res. 2015, 46:37.
-
Gimeno M, Darwich L, Diaz I, de la Torre E, Pujols J, Martín M, Inumaru S, Cano E, Domingo M, Montoya M, Mateu E. Cytokine profiles and phenotype regulation of antigen presenting cells by genotype-I porcine reproductive and respiratory syndrome virus isolates. Vet Res. 2011, 18:42:9.
-
Goldberg TL1, Lowe JF, Milburn SM, Firkins LD. Quasispecies variation of porcine reproductive and respiratory syndrome virus during natural infection. Virology. 2003, 317:197-207.
-
Halbur P, Bush E. Update on abortion storms and sow mortality. Swine Health Prod. 1997, 5:73.
-
Han J, Wang Y, Faaberg KS. Complete genome analysis of RFLP 184 isolates of porcine reproductive and respiratory syndrome virus. Virus Res. 2006, 122:175-82.
-
Virus taxonomy: classification and nomenclature of viruses: Ninth Report of the International Committee on Taxonomy of Viruses. Ed: King AMQ, Adams MJ, Carstens EB, Lefkowitz EJ. Elsevier Academic Press. 2012.
-
Johnson CR, Griggs TF, Gnanandarajah J, Murtaugh MP. Novel structural protein in porcine reproductive and respiratory syndrome virus encoded by an alternative ORF5 present in all arteriviruses. J Gen Virol. 2011, 92:1107-16.
-
Le Gall A, Albina E, Magar R, Gauthier JP. Antigenic variability of porcine reproductive and respiratory syndrome (PRRS) virus isolates. Influence of virus passage in pig. Vet Res. 1997, 28:247-57.
-
Kapur V, Elam MR, Pawlovich TM, Murtaugh MP. Genetic variation in porcine reproductive and respiratory syndrome virus isolates in the midwestern United States. J Gen Virol. 1996, 77:1271-6.
-
Kuhn JH, Lauck M, Bailey AL, Shchetinin AM, Vishnevskaya TV, Bào Y, Ng TF, LeBreton M, Schneider BS, Gillis A, Tamoufe U, Diffo Jle D, Takuo JM, Kondov NO, Coffey LL, Wolfe ND, Delwart E, Clawson AN, Postnikova E, Bollinger L, Lackemeyer MG, Radoshitzky SR, Palacios G, Wada J, Shevtsova ZV, Jahrling PB, Lapin BA, Deriabin PG, Dunowska M, Alkhovsky SV, Rogers J, Friedrich TC, O’Connor DH, Goldberg TL. Reorganization and expansion of the nidoviral family Arteriviridae. Arch Virol. 2016, 161:755-68.
-
Lee C, Yoo D. The small envelope protein of porcine reproductive and respiratory syndrome virus possesses ion channel protein-like properties. Virology. 2006, 355:30-43.
-
Martelli P, Cordioli P, Fallacara F, Gozio S, Terreni M, Cavirani S. A follow up study of recurrent acute PRRS (Atypical PRRS-SAMS) and genetic variations of ORF5. In Proceedings of the 4th International Symposium on Emerging and Re-emerging Pig Diseases, 75-76, 2003, Italy.
-
Martín-Valls GE, Kvisgaard LK, Tello M, Darwich L, Cortey M, Burgara-Estrella AJ, Hernández J, Larsen LE, Mateu E. Analysis of ORF5 and full-length genome sequences of porcine reproductive and respiratory syndrome virus isolates of genotypes 1 and 2 retrieved worldwide provides evidence that recombination is a common phenomenon and may produce mosaic isolates. J Virol. 2014, 88:3170-81.
-
Mengeling WL, Lager KM, Vorwald AC. Clinical consequences of exposing pregnant gilts to strains of porcine reproductive and respiratory syndrome (PRRS) virus isolated from field cases of “atypical” PRRS. Am J Vet Res. 1998, 59:1540–4.
-
Meulenberg JJ, Petersen-den Besten A, De Kluyver EP, Moormann RJ, Schaaper WM, Wensvoort G. Characterization of proteins encoded by ORFs 2 to 7 of Lelystad virus. Virology. 1995, 206:155-63.
-
Meulenberg JJ, Petersen den Besten A, de Kluyver E, van Nieuwstadt A, Wensvoort G, Moormann RJ. Molecular characterization of Lelystad virus. Vet Microbiol. 1997, 55:197-202.
-
Morgan SB, Frossard JP, Pallares FJ, Gough J, Stadejek T, Graham SP, Steinbach F, Drew TW, Salguero FJ. Pathology and virus distribution in the lung and lymphoid tissues of pigs experimentally inoculated with three distinct type 1 prrs virus isolates of varying pathogenicity. Transbound Emerg Dis. 2014. doi: 10.1111/tbed.12272.
-
Morgan SB, Graham SP, Salguero FJ, Sánchez Cordón PJ, Mokhtar H, Rebel JM, Weesendorp E, Bodman-Smith KB, Steinbach F, Frossard JP. Increased pathogenicity of European porcine reproductive and respiratory syndrome virus is associated with enhanced adaptive responses and viral clearance. Vet Microbiol. 2013, 163: 13-22.
-
Murtaugh MP, Elam MR, Kakach LT. Comparison of the structural protein coding sequences of the VR-2332 and Lelystad virus strains of the PRRS virus. Arch Virol. 1995, 140:1451-60.
-
Murtaugh MP, Yuan S, Faaberg KS. Appearance of novel PRRSV isolates by recombination in the natural environment. Adv Exp Med Biol. 2001, 494:31-6.
-
Murtaugh MP, Stadejek T, Abrahante JE, Lam TT, Leung FC. The ever-expanding diversity of porcine reproductive and respiratory syndrome virus. Virus Res. 2010, 154:18-30.
-
Music N, Gagnon CA. The role of porcine reproductive and respiratory syndrome (PRRS) virus structural and non-structural proteins in virus pathogenesis. Anim Health Res Rev. 2010, 11:135-63.
-
Oleksiewicz MB, Stadejek T, Maćkiewicz Z, Porowski M, Pejsak Z. Discriminating between serological responses to European-genotype live vaccine and European-genotype field strains of porcine reproductive and respiratory syndrome virus (PRRSV) by peptide ELISA. J Virol Methods. 2005, 129:134-44.
-
Rowland RR, Steffen M, Ackerman T, Benfield DA. The evolution of porcine reproductive and respiratory syndrome virus: quasispecies and emergence of a virus subpopulation during infection of pigs with VR-2332. Virology. 1999, 259:262-6.
-
Shi M, Lam TT, Hon CC, Hui RK, Faaberg KS, Wennblom T, Murtaugh MP, Stadejek T, Leung FC. Molecular epidemiology of PRRSV: a phylogenetic perspective. Virus Res. 2010, 154:7-17.
-
Sinn LJ, Zieglowski L, Koinig H, Lamp B, Jansko B, Mößlacher G, Riedel C, Hennig-Pauka I, Rümenapf T. Characterization of two Austrian porcine reproductive and respiratory syndrome virus (PRRSV) field isolates reveals relationship to East Asian strains. Vet Res. 2016, 47:17.
-
Snijder EJ, Meulenberg JJ. The molecular biology of arteriviruses. J Gen Virol. 1998, 79:961-79. Snijder EJ, Dobbe JC, Spaan WJ. Heterodimerization of the two major proteins is essential for arterivirus infectivity. J Virol. 2003, 77:97-104. Snijder EJ, Kikkert M, Fang Y. Arterivirus molecular biology and pathogenesis. J Gen Virol. 2013, 94:2141-63.
-
Spilman MS, Welbon C, Nelson E, Dokland T. Cryo-electron tomography of porcine reproductive and respiratory syndrome virus: organization of the nucleocapsid. J Gen Virol. 2009, 90:527-35.
-
Stadejek T, Oleksiewicz MB, Potapchuk D, Podgórska K. Porcine reproductive and respiratory syndrome virus strains of exceptional diversity in Eastern Europe support the definition of new genetic subtypes. J Gen Virol. 2006, 87:1835-41.
-
Stadejek T, Stankevicius A, Murtaugh MP, Oleksiewicz MB. Molecular evolution of PRRSV in Europe: current state of play. Vet Microbiol. 2013, 165:21-8.
-
Sun L, Li Y, Liu R, Wang X, Gao F, Lin T, Huang T, Yao H, Tong G, Fan H, Wei Z, Yuan S. Porcine reproductive and respiratory syndrome virus ORF5a protein is essential for virus viability. Virus Res. 2013, 171:178-85.
-
Tian D, Wei Z, Zevenhoven-Dobbe JC, Liu R, Tong G, Snijder EJ, Yuan S. Arterivirus minor envelope proteins are a major determinant of viral tropism in cell culture. J Virol. 2012, 86:3701-12.
-
Truong HM, Lu Z, Kutish GF, Galeota J, Osorio FA, Pattnaik AK. A highly pathogenic porcine reproductive and respiratory syndrome virus generated from an infectious cDNA clone retains the in vivo virulence and transmissibility properties of the parental virus. Virology. 2004, 325:308–19.
-
Van Vugt JJ, Storgaard T, Oleksiewicz MB, Bøtner A. High frequency RNA recombination in porcine reproductive and respiratory syndrome virus occurs preferentially between parental sequences with high similarity. J Gen Virol. 2001, 82:2615-20.
-
Wang X, Marthaler D, Rovira A, Rossow S, Murtaugh MP. Emergence of a virulent porcine reproductive and respiratory syndrome virus in vaccinated herds in the United States. Virus Res. 2015, 210:34-41.
-
Weiland E, Wieczorek-Krohmer M, Kohl D, Conzelmann KK, Weiland F. Monoclonal antibodies to the GP5 of porcine reproductive and respiratory syndrome virus are more effective in virus neutralization than monoclonal antibodies to the GP4. Vet Microbiol. 1999, 66:171-86.
-
Wissink EH, Kroese MV, van Wijk HA, Rijsewijk FA, Meulenberg JJ, Rottier PJ. Envelope protein requirements for the assembly of infectious virions of porcine reproductive and respiratory syndrome virus. J Virol. 2005, 79:12495-506.
-
Zhao K, Ye C, Chang XB, Jiang CG, Wang SJ, Cai XH, Tong GZ, Tian ZJ, Shi M, An TQ. Importation and Recombination Are Responsible for the Latest Emergence of Highly Pathogenic porcine reproductive and respiratory syndrome virus in China. J Virol. 2015, 89:10712-6.
-
Zimmerman JJ, Benfield DA, Dee SA, Murtaugh MP, Stadejek T, Stevenson GW, Torremorell M. Porcine reproductive and respiratory syndrome virus (porcine arterivirus). In: 10th ed. Diseases of swine, Ed. Wiley-Blackwell. 2012, 31:463-86.